Résistance des Bactéries aux Antibiotiques |
L'Antibiorésistance Tue plus de 1 Million de Personnes par An |
T.A. - SCIENCE & VIE N°1258 > Juillet > 2022 |
1,27 Millions de Morts d'Infections Bactériennes Résistantes aux Antibiotiques en 2019 |
LE MONDE > 26 Janvier > 2022 |
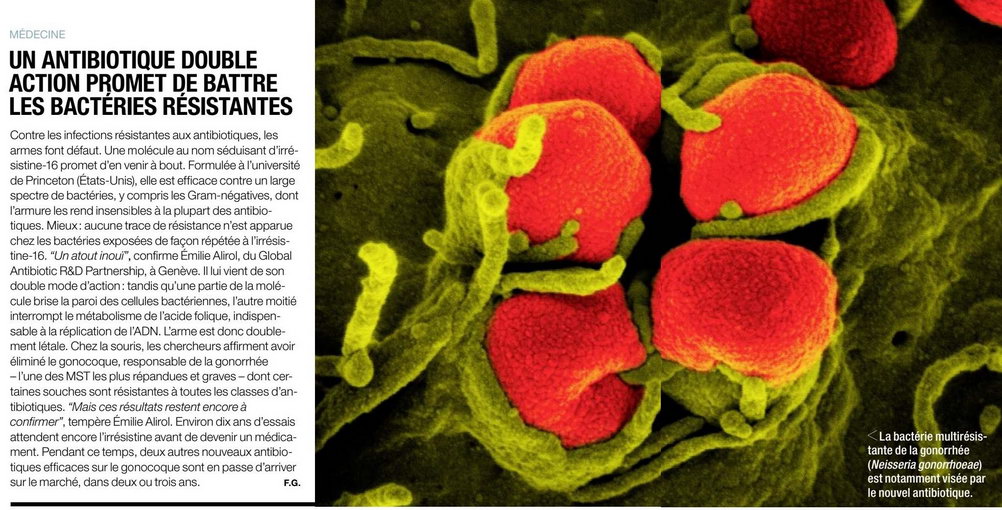
Un Antibiotique Double action promet de battre les Bactéries Résistantes |
F.G. - SCIENCE & VIE N°1235 > Août > 2020 |
Antibiorésistance : et Si on s'Inspirait des Fourmis |
A.A. - LA RECHECHE N°554 > Décembre > 2019 |
Antibiorésistance : impact en Hausse en Europe |
POUR LA SCIENCE N°495 > Janvier > 2019 |
Les Surfeurs plus Exposés aux Bactéries Résistantes |
ÇA M'INTÉRESSE HS N°9 > Avril-Mai > 2018 |
La Plante Tueuse de Bactéries : Faux poivrier |
A.-R.M. - NATIONAL GEOGRAPHIC N°217 > Octobre > 2017 |
La Résistance aux Antibiotiques Remonte à la Préhistoire |
 Après des années de recherches sur l'ADN ancien du pergélisol (sous-sol gelé) des territoires du Yukon daté d'environ 30.000 ans, une équipe de chercheurs franco-canadienne a développé des méthodes permettant d'isoler des gènes particuliers de paléo-bactéries au Centre d'ADN ancien de l'université McMaster (Canada) et au Service de Systématique Moléculaire du Muséum national d'Histoire naturelle.
Après des années de recherches sur l'ADN ancien du pergélisol (sous-sol gelé) des territoires du Yukon daté d'environ 30.000 ans, une équipe de chercheurs franco-canadienne a développé des méthodes permettant d'isoler des gènes particuliers de paléo-bactéries au Centre d'ADN ancien de l'université McMaster (Canada) et au Service de Systématique Moléculaire du Muséum national d'Histoire naturelle.
En utilisant les techniques les plus pointues dans le domaine de la biologie moléculaire, les chercheurs ont d'abord mis au point une armada de tests visant à authentifier l'origine des ADN anciens recouvrés dans ces carottes de sol gelé. Des méthodes de code-barres ADN ont ainsi été développées pour extraire des petites séquences d'ADN ancien d'organismes variés : plantes, vertébrés, et donc bactéries. Ces pergélisols ont révélé une grande diversité biologique parmi ces ADN anciens : des séquences de mammouths, de chevaux, de bisons ainsi que de plantes qui n'ont été retrouvés dans cette localité que dans la dernière période glaciaire du pléistocène (-130.000 à -11.000 ans). De manière inattendue, les chercheurs ont découvert des gènes bactériens de résistance aux antibiotiques parmi ces échantillons d'ADN ancien.
Les chercheurs se sont concentrés plus particulièrement sur une séquence du gène de la résistance à la vancomycine, un antibiotique très puissant de la famille des glycopeptides, généralement utilisé en dernier recours dans le domaine clinique. Or, des résistances à cet antibiotique ont émergé dans les années 1980, et causent aujourd'hui encore dans le monde entier des flambée d'infections nosocomiales. La présence d'homologues de ce gène, c'est-à-dire de gènes très proches, a été identifiée indépendamment dans le laboratoire d'ADN ancien canadien et l'unité "Eco-Anthropologie et ethnobiologie" (MNHN/CNRS), à partir de différents extraits d'ADN issus des mêmes carottes de pergélisol. Il a ainsi été démontré que ces gènes appartenaient à des bactéries contemporaines des mammouths et non à des bactéries modernes dont ils sont génétiquement proches mais présentent également des mutations singulières. L'équipe a ensuite recréé le produit du gène en laboratoire et démontré que les protéines obtenues avaient une activité et une structure similaires à celles existant aujourd'hui. C'est seulement la deuxième fois qu'une protéine dérivée d'une séquence d'ADN ancien est synthétisée dans un laboratoire. Cette découverte apporte un nouvel éclairage sur la compréhension de la résistance aux antibiotiques en démontrant l'ancienneté et le potentiel adaptatif de ces gènes. L'objectif des chercheurs est désormais de poursuivre leurs travaux dans le pergélisol remontant à 1 million d'années.
SCIENCE MAGAZINE N°33 > Février-Mars-Avril > 2012 |
La Résistance des Bactéries aux Antibiotiques se Répand comme une Épidémie |
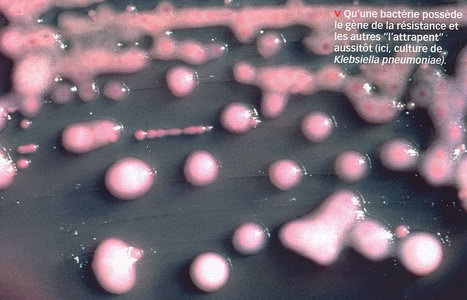 "Des bactéries sournoises qui laissent craindre une pandémie inédite", voilà comment Jean-Marc Rolain, spécialiste de la résistance aux antibiotiques à l'hôpital de la Timone, à Marseille, qualifie les bactéries ultra résistantes découvertes voilà un an à peine à New Delhi (Inde).
"Des bactéries sournoises qui laissent craindre une pandémie inédite", voilà comment Jean-Marc Rolain, spécialiste de la résistance aux antibiotiques à l'hôpital de la Timone, à Marseille, qualifie les bactéries ultra résistantes découvertes voilà un an à peine à New Delhi (Inde).
Car on en est désormais certain : tous les continents sont touchés. Ces bactéries, détectées au total chez 200 patients - la grande majorité ayant séjourné auparavant dans un hôpital en Inde ou au Pakistan - sont potentiellement résistantes à tous les antibiotiques connus.
UNE ARME GÉNÉTIQUE
Pourtant, il ne s'agit pas d'un "nouveau" microbe, mais d'un gène de résistance appelé NDM-1 qui se retrouve chez des bactéries banales mais capables de provoquer des infections, comme Klebsiella pneumoniae ou Escherichia coli. "Ce gène leur permet de détruire les antibiotiques utilisés à l'hôpital en dernier recours, explique Jean-Marc Rolain. Elles deviennent résistantes à tous les antibiotiques, sauf un, la colistine, peu utilisée car très toxique. De plus, certaines espèces de bactéries résistent naturellement à la colistine. Avec le gène NDM-1, elles deviennent donc invincibles".
Or, cette arme génétique se répand comme une épidémie. "Le gène NDM-1 est localisé sur un élément d'ADN mobile, le plasmide, transmis d'une bactérie à une autre. Ainsi, si une seule bactérie sur une population bactérienne de 10 millions possède ce gène, toutes peuvent 'l'attraper' en moins de 24 heures", poursuit le spécialiste. Pis, ce gène est capable de "sauter" d'espèce en espèce de façon explosive. Il a déjà été trouvé chez une dizaine d'espèces de bactéries. "En France, on recherche systématiquement les bactéries NDM-1 chez les patients rapatriés d'un hôpital étranger et on isole ceux qui y sont positifs. Mais il est impossible de savoir combien de personnes sont porteuses de ces bactéries sans pour autant être malades", s'inquiète Jean-Marc Rolain.
M.Co. - SCIENCE & VIE > Décembre > 2010 |
C'est par un Subterfuge Métabolique que les Bactéries se jouent des Antibiotiques |
 On savait les bactéries capables de s'adapter rapidement aux antibiotiques, au point que certaines molécules sont devenues inefficaces pour traiter certaines infections.
On savait les bactéries capables de s'adapter rapidement aux antibiotiques, au point que certaines molécules sont devenues inefficaces pour traiter certaines infections.
Des chercheurs associés de l'Inserm, l'université Paris-V, l'Inra, l'Institut Pasteur et le CNRS ont identifié l'une de leurs stratégies, qui consiste à détourner les acides gras présents dans le sang humain pour construire leur membrane. Certains antibiotiques sont justement censés empêcher les bactéries de fabriquer leurs propres acides gras.
Pour Claire Poyart, l'une des auteurs de l'étude, "il s'agit même d'un véritable parasitisme métabolique". Confirmée in vivo, la découverte remet en question l'utilisation des antibiotiques ciblant la fabrication des acides gras, des médicaments pourtant prometteurs qui avaient fait la preuve de leur efficacité in vitro.
SCIENCE & VIE > Mars > 2007 |
La Résistance aux Antibiotiques est Finalement bien Plus Vaste |
 On savait déjà que les bactéries du sol développaient des résistances aux antibiotiques. Mais l'ampleur du phénomène était largement sous-estimée, comme vient de le démontrer le biologiste Canadien Gerard Wright, de l'université McMaster.
On savait déjà que les bactéries du sol développaient des résistances aux antibiotiques. Mais l'ampleur du phénomène était largement sous-estimée, comme vient de le démontrer le biologiste Canadien Gerard Wright, de l'université McMaster.
Pour ce faire, le chercheur a extrait de terrains agricoles, forestiers ou urbains 480 bactéries différentes (du genre Streptomyces ->). Il les a confrontées à 21 antibiotiques naturels ou synthétiques. Résultat : chaque bactérie est en moyenne résistante à sept ou huit antibiotiques. Une résistance qui pourrait être d'origine naturelle : la plupart des antibiotiques sont fabriqués à partir de bactéries dotées de mécanismes d'auto-défense contre leurs propres toxiques. Reste que des résistances ont également pu être engendrées par la sélection de microbes confrontés dans la nature à des molécules médicamenteuses.
Certes, les bactéries du sol ne transmettraient pas directement leurs "capacités de résistance" aux pathogènes responsables de maladies chez l'homme. Mais, d'après Gerard Wright, ce transfert a certainement déjà eu lieu, via des souches intermédiaires. "La surveillance de ce réservoir microbien permettra de faciliter le développement d'une nouvelle génération d'antibiotiques", conclut le chercheur.
F.T. - SCIENCE & VIE > Mars > 2006 |