Bioprocesseur - Paré pour le Calcul Moléculaire |
 Calculer plus vite, avec moins d'énergie et moins d'encombrement, ce rêve d'informaticien, la biologie a cru le réaliser dans les années 1990 grâce au nombre infini de combinaisons qu'offrent les manipulations de l'ADN et la capacité des cellules à répondre à quantité de messages simultanés. Aujourd'hui, c'est finalement un autre usage qui s'impose : l'intervention au cour même des cellules de dispositifs biologiques synthétiques.
Calculer plus vite, avec moins d'énergie et moins d'encombrement, ce rêve d'informaticien, la biologie a cru le réaliser dans les années 1990 grâce au nombre infini de combinaisons qu'offrent les manipulations de l'ADN et la capacité des cellules à répondre à quantité de messages simultanés. Aujourd'hui, c'est finalement un autre usage qui s'impose : l'intervention au cour même des cellules de dispositifs biologiques synthétiques.
Certes, il en faudrait mille milliards pour remplir un seul tube à essais. Mais son pouvoir est démesuré ! "Qui" ? C'est un bioprocesseur entièrement réalisé à partir d'acide ribonucléique (ARN), soit l'une des briques les plus élémentaires du vivant. Mesurant à peine quelques nanomètres, il peut être implanté dans une cellule vivante, afin d'y détecter la présence de substances, réagir, informer sur l'état de la cellule...
Mais au-delà de cet espionnage biologique, l'appareil fonctionne surtout comme un vrai ordinateur moléculaire, capable d'effectuer des calculs à partir des informations captées à l'intérieur de la cellule et d'en délivrer un résultat. Du coup, le système est en mesure d'intervenir dans le fonctionnement de la cellule pour y déclencher des réactions chimiques, bloquer un processus ou en activer d'autres... Une première !
Cette étonnante biomachine est l'ouvre d'une ingénieure chimiste américaine, Christina Smolke, qui l'a conçue et fabriquée au California Institute of Technology (Caltech), à Pasadena. Et elle vient d'en démontrer l'efficacité : inséré dans une cellule vivante de levure (Saccharomyces cerevisiae), son bioprocesseur peut recevoir des signaux (la présence ou l'absence de molécules simples) et y répondre par un signal fluorescent !  À ce jour, il s'agit du résultat le plus spectaculaire d'une toute nouvelle science : la biologie synthétique. (Enregistrement de la fluorescence des cellules qui signale la réponse du bioprocesseur qu'elles hébergent. ->)
À ce jour, il s'agit du résultat le plus spectaculaire d'une toute nouvelle science : la biologie synthétique. (Enregistrement de la fluorescence des cellules qui signale la réponse du bioprocesseur qu'elles hébergent. ->)
AGIR AU COUR DE LA CELLULE
Ses missions ? Construire des capteurs, des circuits logiques, des usines à protéines... à l'intérieur de cellules vivantes afin de les soigner, leur faire produire des substances ou simplement observer ce qui s'y passe sans en perturber le fonctionnement naturel. "Après les longues années de décryptage des gènes par les biologistes, le temps de leur manipulation par des ingénieurs, des logiciens voire des physiciens est venu", commente Alfonso Jaramillo, ex-physicien qui enseigne à présent la biologie synthétique à l'Ecole polytechnique.
Historiquement, la création du premier ordinateur "génétique" par Leonard Adleman, de l'University of Southem Califomia, date de 1994. Le projet était alors de voir si la biologie pouvait permettre de miniaturiser les processeurs en profitant des capacités de calcul du vivant. En particulier, on pensait que l'ADN, vu sa taille insignifiante et les possibilités de combinaisons qu'offrent sa structure et ses quatre éléments de base, remplacerait un jour les tout premiers Pentium sortant des labos d'Intel. Et de fait, dans un tube à essais, un programme écrit sur une molécule d'ADN parvint à résoudre le "problème du voyageur de commerce" (calculer le trajet le plus court d'une tournée passant par plusieurs villes), connu pour sa complexité mathématique. "Cependant, ce n'est pas tant leur potentiel de taille ou de vitesse de calcul qui rend les bioprocesseurs intéressants, mais le fait qu'ils parlent le langage du vivant. Et c'est donc dans la cellule qu'ils trouveront leurs applications", plaide Christina Smolke.
Le mouvement s'est concrétisé en 2004 avec l'élaboration, par Ehud Shapiro du Weizmann Institute, en Israël, d'une biomachine formée d'un double brin d'ADN capable de détecter un marqueur du cancer et de produire, en réponse, un médicament anticancéreux. Un grand succès, obtenu en éprouvette. Depuis, d'autres "biomachines" du même type ont été créées, mais celle de Christina Smolke et de son doctorant en chimie Maung Nyan Win, annoncée cet automne dans la revue Science, est la plus aboutie. Grâce, notamment, au choix du matériau. Cette fois, ce n'est plus de l'ADN qui supporte le processeur, mais un ingénieux assemblage de courtes séquences d'ARN. Une molécule étonnante, dont la composition est proche de celle de l'ADN, sur laquelle de nombreux "bio-ingénieurs" fondent de grands espoirs. "L'ARN est le matériau idéal pour l'ingénierie biomoléculaire car ses fonctions sont multiples", confirme Eric Westhof, directeur de l'unité architecture et réactivité de l'ARN du CNRS, à l'université Louis-Pasteur à Strasbourg.
La fonction la plus connue de l'ARN est de véhiculer l'information inscrite dans l'ADN vers l'extérieur du noyau, où sont fabriquées les protéines. Il est donc possible d'en assembler les bases pour qu'il fabrique toutes sortes de protéines. Mais l'ARN peut aussi se présenter sous une forme particulière, appelée "aptamère". Il agit alors comme un capteur capable de se lier à une molécule cible. Sous forme dite "ribozyme", il a cette fois une action mécanique. Ainsi, le ribozyme dit "à tête de marteau" agit comme une paire de ciseaux qui coupe en deux la molécule sur laquelle il est fixé.
Or, ces différentes formes que prend l'ARN peuvent être réunies en séquences consécutives pour créer une unique molécule d'ARN aux formes bizarres et aux fonctions multiples. On obtient alors un "transmetteur d'information", en profitant du fait que chaque action d'une de ses séquences modifie la structure de la molécule d'ARN dans l'espace. En clair : tout réarrangement du brin peut être interprété comme un signal qui déclenche à son tour une nouvelle action.
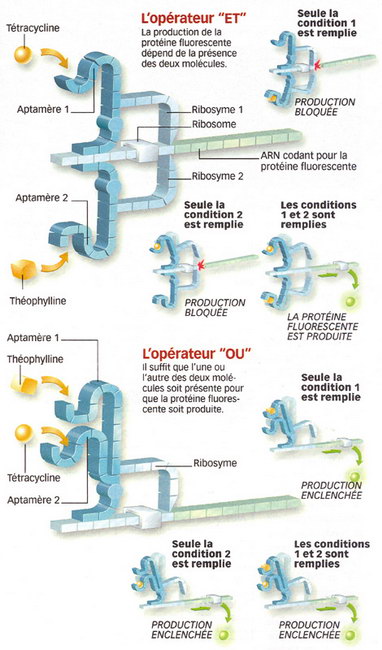 Les montages d'ARN réalisent des opérations logiques élémentaires (infographie ->)
Les montages d'ARN réalisent des opérations logiques élémentaires (infographie ->)
Une machine biomoléculaire est assemblée à partir de 3 ARN : l'un codant pour une protéine fluorescente, un autre, via 2 aptamères servant de récepteurs et enclenchant, chacun, un troisième ARN (ribozyme) capable de couper la séquence codante pour la protéine fluorescente du brin d'ARN. lci, selon la présence ou l'absence de molécules-signal, l'assemblage remplit la fonction d'un opérateur logique soit "ET", soit "OU".
PROGRAMMATION DE "CALCULS"
Concrètement, le bioprocesseur de la chercheuse du Caltech se présente sous la forme d'une unique molécule d'ARN. Elle comprend une séquence codante pour une protéine fluorescente, à laquelle ont été fixés deux aptamères différents, chacun lié à un ribozyme "à tête de marteau" (->). Une fois insérée dans la cellule (par osmose, une méthode classique de biologie moléculaire), la biomachine de Christina Smolke fonctionne comme un véritable ordinateur moléculaire, n'attendant plus qu'un signal d'entrée. À savoir la présence ou l'absence des deux molécules cibles des aptamères, en l'occurrence la tétracycline et la théophylline. S'ils trouvent leurs cibles dans leur environnement, les aptamères s'y fixent, ce qui modifie leur arrangement dans l'espace, et donc celui de la molécule d'ARN tout entière. Commence alors la partie "calcul logique" du système : dans sa configuration de base, les ribozymes sont conçus pour sectionner l'ARN sur lequel ils sont accrochés (le brin coupé en deux est alors dégradé par la cellule). Il suffit qu'un seul ribozyme conserve sa place originelle sur la molécule pour que les ciseaux fassent effet. Mais le changement de conformation de la molécule d'ARN dû aux mouvements des aptamères désactive chacun des ribozymes. Aucun des ciseaux ne fonctionnant plus, la molécule d'ARN reste intacte. La protéine fluorescente est produite. C'est le signal de sortie du processeur. En bref, un opérateur logique "ET" vient d'être créé : la présence de tétracycline ET de théophylline a provoqué un signal de sortie !
"La force d'un tel outil, c'est qu'il est très malléable : selon l'emplacement des aptamères et des ribozymes dans la molécule, le processeur réagit différemment", commente Alfonso Jaramillo. L'opérateur logique "OU" peut être ainsi fabriqué en combinant les séquences d'ARN de façon que la présence d'une seule des deux molécules cibles dans la cellule bloque les ribozymes et permette la production de la protéine fluorescente. Les opérateurs "NON-ET" et "NON-OU" sont aussi programmables. Grâce à une coopération mondiale initiée parle MIT (encadré), une trentaine d'aptamères "naturels" ont déjà été recensés dans divers labos. Ce sont autant de signaux d'entrée possibles pour un bioprocesseur. Quant aux signaux de sortie, il peut s'agir de toutes sortes de protéines aux fonctions intéressantes. Il ne reste plus qu'à assembler les briques du mécano pour construire des biomachines de plus en plus sophistiquées ! Sachant que, en théorie, de tels engins pourraient fonctionner dans n'importe quelle cellule vivante. "Nous l'avons inséré dans des cellules de levure, mais nous aurions pu utiliser des cellules de mammifères ou des bactéries", affirme Christina Smolke.
Antoine Danchin, directeur de l'unité génétique des génomes bactériens de l'Institut Pasteur, reste toutefois sceptique : "La démonstration n'a été réalisée que sur un organisme unicellulaire ; or, l'ARN est très versatile. On dit que l'ARN est une molécule qui respire, car elle s'ouvre et se ferme sans cesse. Elle est loin d'être aussi rigide que la logique binaire le réclame, et je doute qu'elle agisse comme espéré chez un mammifère, par exemple."
L'INGENIERIE OPEN SOURCE En 2004, le Massachusetts Institute of Technology (MID s'est lancé dans la création d'un catalogue de "biobriques" élémentaires dont le but est de recenser toute séquence génétique aux fonctions connues. Le catalogue et son recueil de procédures de fabrication sont mis à la disposition des chercheurs du monde entier, selon le principe de "l'open source". Les scientifiques y puisent des matériaux de base, dont des aptamères ou des ribozymes, pour des constructions qui peuvent aller du programme génétique le plus simple à la création de cellules complètement artificielles. Pour stimuler la création de nouvelles biobriques et le design de toutes sortes de biomachines, un concours annuel baptisé IGEM (International Genetically Engineered Machinery) est ouvert aux étudiants. Chaque équipe doit améliorer une cellule vivante en lui conférant de nouvelles fonctions. Ont notamment été créés ces dernières années : une bactérie mangeuse de graisse, un microbe photographe ou encore une levure imprimante ! Cette année, l'équipe slovène a gagné le trophée avec un vaccin contre la bactérie Helicobacter pylori responsable d'ulcères et de cancers de l'estomac. C'est un des rares projets qui devrait perdurer au-delà de la compétition tant les résultats préliminaires sont prometteurs. |
DE LA LEVURE À L'HOMME ?
On devrait en avoir le cour net prochainement puisque Christina Smolke annonce sa collaboration avec un centre de recherche contre le cancer pour tester son bioprocesseur sur des animaux. Cette fois, le signal d'entrée sera un biomarqueur du cancer. "On imagine une thérapie moléculaire qui traitera les cellules malades et laissera tranquille les cellules saines", avance l'ingénieure. Dans un autre domaine, des "biomachines" pourront détecter et traiter les polluants dans des plantes contaminées. À plus long terme, les cellules vivantes pourraient même produire des biocarburants ou d'autres produits de chimie fine... Qui dit mieux ?
I.C. - SCIENCE & VIE > Janvier > 2009 |